微生物研究工作中,我们常常需要检测某些特定微生物的数量,比如特定种(Species)或特定属(Genus),甚至特定科(Family)微生物的存在和数量。
采用qPCR及ddPCR技术,理论上,可以进行特定微生物菌群的定量检测,但要求其对应的qPCR及ddPCR引物、探针,可以特异性检测到该类微生物菌群,而与非该类的微生物不产生非特异性反应。
众所周知,微生物具有较高的变异率,因此哪怕是同一个种(Species)水平内的微生物菌株,菌株间基因组的整体差异度有可能会高达30%以上。能选做特定菌群检测靶点的基因,需要在所有待检测的微生物菌群中具有相当高的同源性,而在其他所有的微生物菌群中,其序列相似性应尽量低一些。
基于上述原因,能很好设计出满足上述要求的引物、探针的难度较高。微基生物推荐采用的特定微生物菌群特异性引物、探针设计策略,是基于目标微生物全基因组序列来进行排查设计的,不推荐只利用16S,ITS 等一段特定基因序列。基于全基因组序列进行设计,可供筛选的基因靶点大大丰富,更有利于筛选出优秀的特异性引物、探针。但这样一来,大幅增加了工作难度和工作量,微基生物经过潜心研发、实践,在过去5年中,我们已经成功为众多客户设计、验证出上190余种特定微生物菌群的特异性引物、探针,积累了丰富的经验,如您有特定微生物菌群特异性引物、探针的设计需求,欢迎联系我们: 400-660-9270,我们将竭诚帮助您达成实验目标。
下面我们将以产酸克雷伯菌Klebsiella oxytoca 的特异性引物、探针设计,来说明此类引物、探针的设计原理及流程。
示例:
产酸克雷伯菌Klebsiella oxytoca特异性引物探针设计
产酸克雷伯菌链接:https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?mode=Info&id=571
1.在NCBI的基因组Genbank、Refseq数据库中,检索下载产酸克雷伯菌全部已知菌株的基因组序列。
下载了Genbank数据库347个基因组,Refseq数据库275个基因组,共计622个基因组序列。
2.对上述622个已知菌株基因组,提取其CDS序列,没有CDS序列的基因组使用软件prodigal预测CDS;
3.使用软件cdhit对上述所有CDS聚类,并得出每条CDS在各自基因组中的拷贝数信息;
4.筛选在上述622条基因组中都存在,且单拷贝的共有核心基因序列;
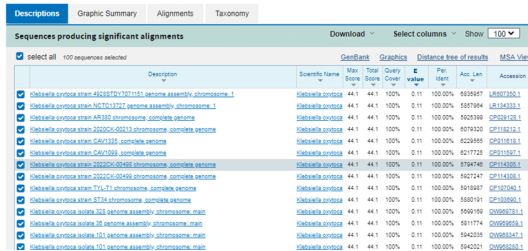
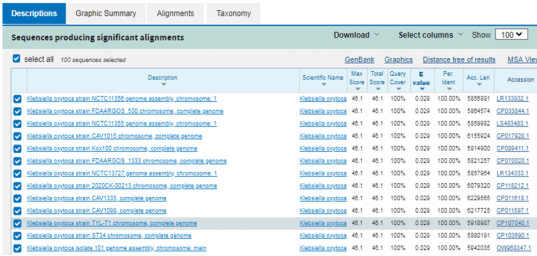
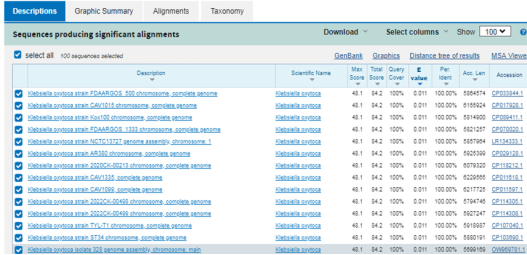
5.将上述共有核心基因序列,分别与NCBI的NT库、Refseq 数据库、检测宿主基因组数据库BLAST比对。
筛选出在NT库和Refseq库中击中不了其他物种,也击中不了宿主基因组的基因序列(下称候选靶标基因序列);用于后继引物、探针的设计。
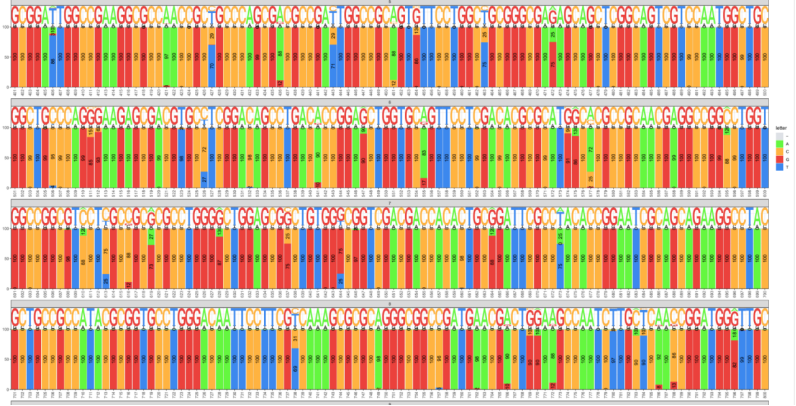
6. 针对每个候选靶标基因序列,调取622个已知菌株基因组中对应的基因序列,利用生信软件统计其每个位置上的序列多样性,选择高保守的序列进行引物和探针设计;
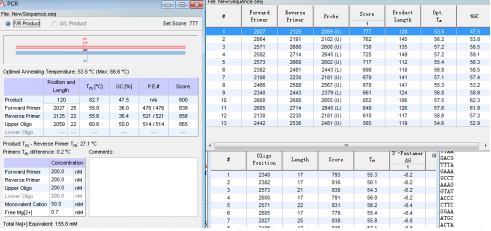
7.采用oligo软件验证引物和探针,查看引物探针的退火温度等信息;
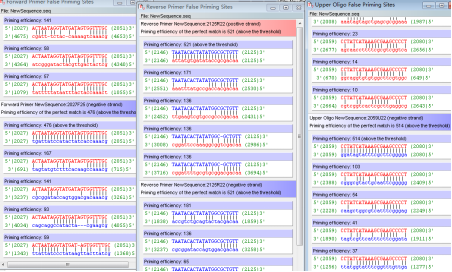
8.将引物、探针与NCBI进行blast比对,查看引物探针的特异性;
上游引物
下游引物
探针序列
9. 合成引物、探针,采用PCR、qPCR、ddPCR验证其扩增特异性、灵敏度、抗宿主基因组干扰性,正式开展特定菌群的绝对定量实验。
9.1引物特异性试验
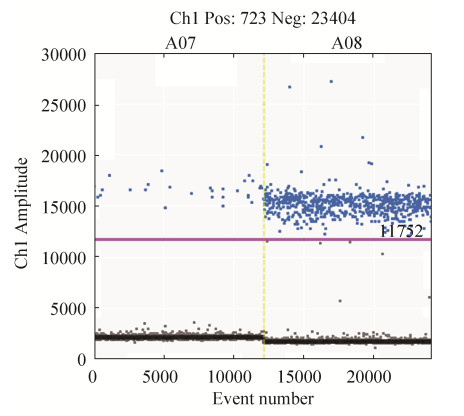
琼脂糖凝胶电泳:引物对2株阳性检测菌株及20株阴性检测菌株扩增结果。
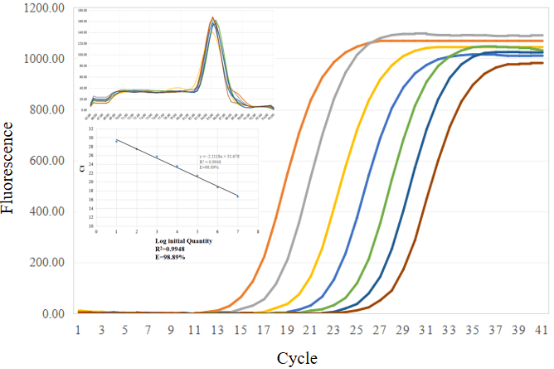
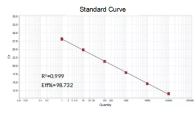
9.2 qPCR扩增灵敏度测试,qPCR扩增特异性测试(溶解曲线)
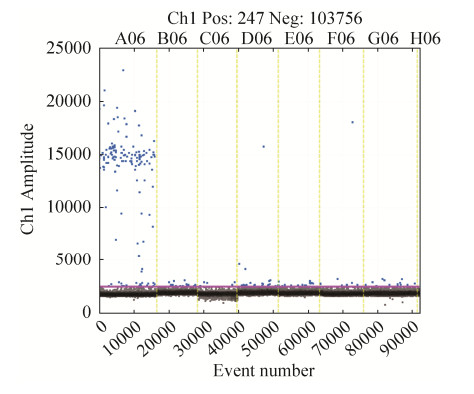
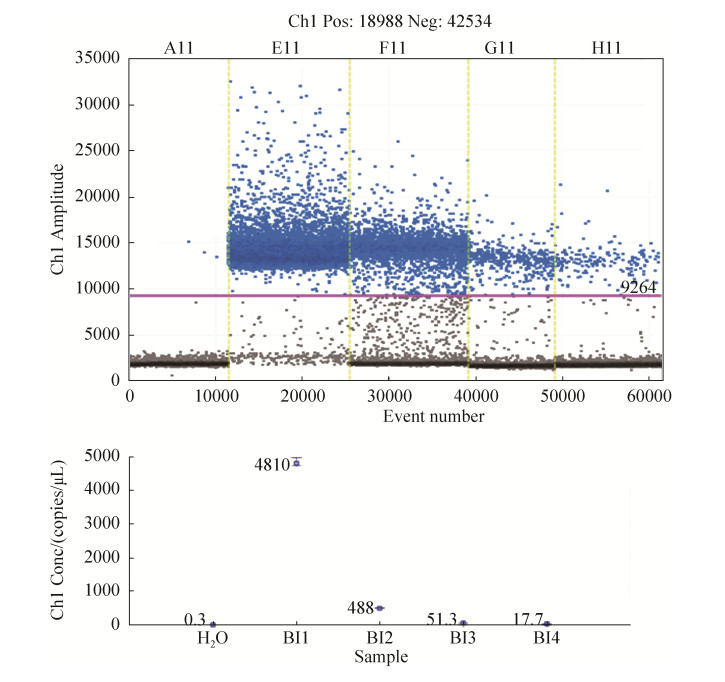
9.3 ddPCR定量验证
引物特异性是决定定量准确度的关键因素之一。引物设计理论结合特异性试验这种方法可以快速有效地筛选出特异性较好的引物。
补充拓展:
1.ChatGTP来答疑:特定微生物qPCR探针引物设计,大致策略?
引物和探针是影响荧光定量PCR成功与否以及其结果是否可靠的关键因素之一。
2.特异性引物与通用引物不同:
以细菌为例,通用引物就是对所有种类的细菌都通用,即用通用引物可以PCR扩增所有细菌的某段DNA。
特异性引物只能用于某种或某类细菌DNA的PCR扩增。
3.引物质量判断
扩增效率熔解曲线引物的扩增效率达到90%-110%,线性相关性R2>0.98即认为扩增效率较好。熔解曲线单峰且通常Tm>80℃即认为扩增特异性较好。
微基生物推出属特异性引物设计服务,能更有效的:
a.鉴定特定物种,以及检测某一微生物群落中是否包含该物种。
b.确定特定物种在整个微生物群落中的丰度。

 微基生物 您自己的微生态研究团队|专注微生态研究与应用
微基生物 您自己的微生态研究团队|专注微生态研究与应用