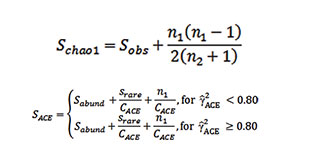>分类学分析
在之前的分析步骤中,已经将序列按照其自身的碱基组成的相似性,分归到各OTU 中。在进行分类学分析时,首先,将每一条优质序列都与SILVA 119数据库进行比对,找出其最相近且可信度达80%以上的种属信息。之后,将每一个OTU 中的所有序列进行类比,找出同一OTU 中的不同序列的最近祖先的种属信息。最后,将得到的结果记录在表格文件中。这样做,可以在保留最可能多的信息量的情况下,确保得出信息的准确性。
使用软件:mothur 例表: 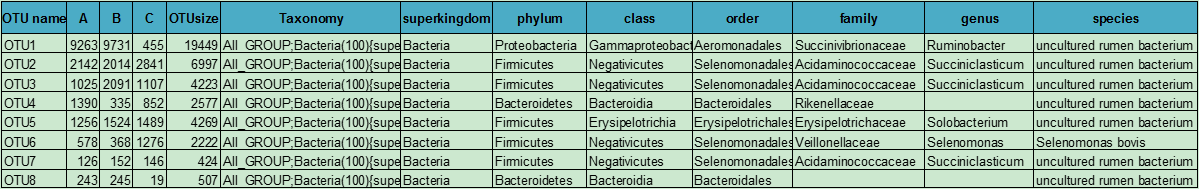 OTU name 为OTU 编号;
OTU name 为OTU 编号;
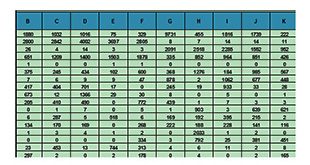
第二列至OTUsize列的前一列为各样本的序列在所有OTU 中的含有情况。例如,第二行第二列的数字代表样品A中有多少序列被划分入OTU1 中。
OTUsize 为该OTU 中所含序列的数量;
Taxonomy列为OTU 对应的种属信息。种属信息按照分类学水平分为多列,我们将物种的门、纲、目、科、属、种的信息进行了分类分析,便于对数据的筛选提取。例如,需要提取所有含有属信息的OTU 的相关信息,可在excel 中选取属这一列,在工具栏的数据项中,点选筛选,查看该列第一行的单元格,在下拉菜单中的文本筛选项下方的区域内,取消选择“空白” ,点确定,即得到所有含有属信息的OTU信息。
注:分类学数据库中会出现一些分类学谱系中的中间等级没有科学名称,以norank 作为标记。分类学比对后根据置信度阈值的筛选,会有某些分类谱系在某一分类级别分值较低,在统计时以Unclassified 标记。
将OTU 综合分类表中的信息按照门、纲、目、科、属、种6 个水平分别提取信息,分别统计各样品在不同分类水平上的菌群组成及丰度。
例表: 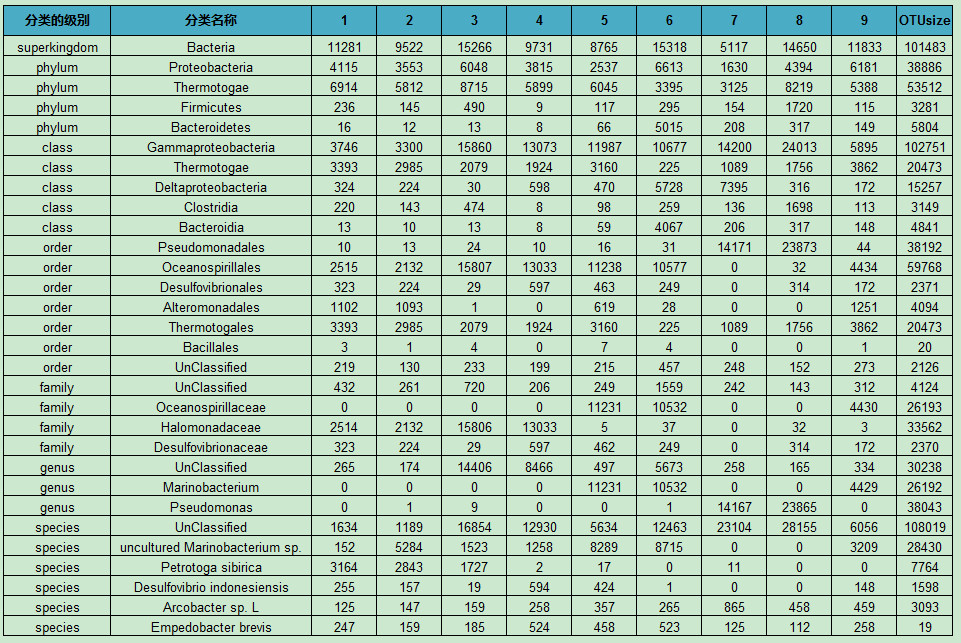
>群落结构组成柱状图
根据分类学分析结果,可以得知一个或多个样品在各分类水平上的分类学比对情况。在结果中,包含了两个信息:
样品中含有何种微生物;
样品中各微生物的序列数,即各微生物的相对丰度。
因此,可以使用统计学的分析方法,观测样品在不同分类水平上的群落结构。将多个样品的群落结构分析放在一起对比时,还可以观测其变化情况。根据研究对象是单个或多个样品,结果可能会以不同方式展示。通常使用较直观的饼图或柱状图等形式呈现。群落结构的分析可在任一分类水平进行。
软件:基于物种分类信息的数据表,利用R 语言工具作图或在EXCLE 中编辑作图。
参考文献:
Lisa Oberauner, Christin Zachow, Stefan Lackner, et al. The ignored diversity: complex bacterial communities in intensive care units revealed by 16S pyrosequencing. SCIENTIFIC REPORTS. 3 :1413 .DOI: 10.1038/srep01413.(2013)
例图: 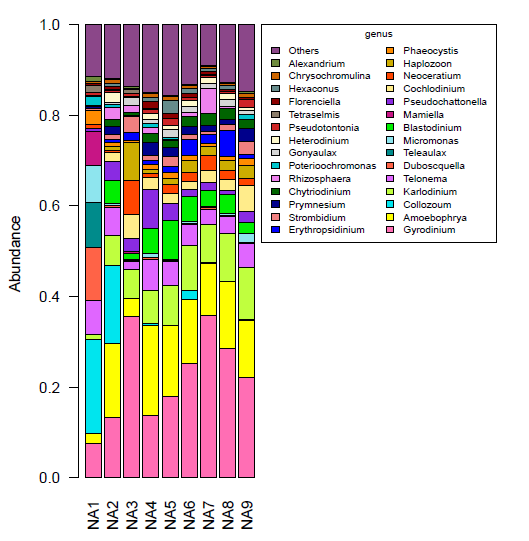
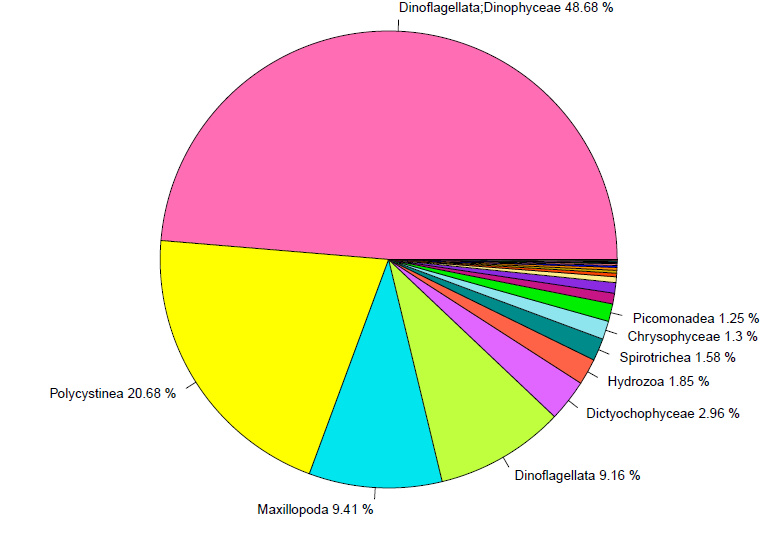 注:为保证视图效果,建议作图时可将丰度极低的部分合并为other 在图中显示
注:为保证视图效果,建议作图时可将丰度极低的部分合并为other 在图中显示
>多样品相似度树与柱状图组合分析
左边是样品间基于群落组成的层次聚类分析(bray-curtis 算法),右边是样品的群落结构柱状图。
参考文献:
Srinivasan S, Hoffman NG, Morgan MT, Matsen FA, Fiedler TL, et al. (2012) Bacterial Communities in Women with Bacterial Vaginosis: High Resolution Phylogenetic Analyses Reveal Relationships of Microbiota to Clinical Criteria. PLoS ONE 7(6): e37818. doi:10.1371/journal.pone.0037818.
例图: 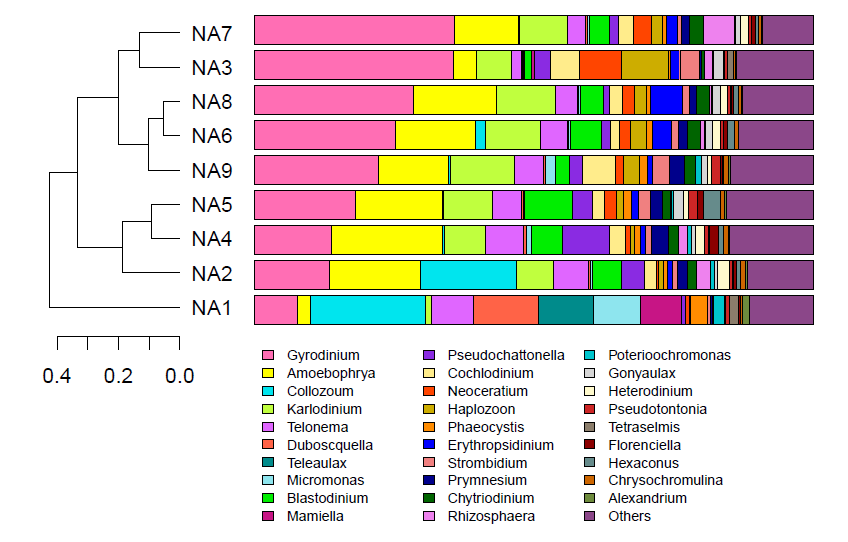

 微基生物 您自己的微生态研究团队|专注微生态研究与应用
微基生物 您自己的微生态研究团队|专注微生态研究与应用