一 背景
肿瘤微生物组学(Tumor Microbiome Studies)是研究肿瘤微环境中微生物群落的科学,它聚焦于研究存在于肿瘤组织微环境中的微生物群落,包括细菌、真菌、病毒以及可能的其他微生物实体。这些微生物与宿主细胞之间的相互作用可能对肿瘤的发生、发展、转移乃至对治疗的响应产生重要影响。
肿瘤微生物组学作为肿瘤研究的新领域,为我们理解肿瘤的复杂性提供了新的视角,并有潜力为癌症的预防、诊断和治疗带来革新性的变化。随着研究的深入,我们期待这一领域能够揭示更多关于肿瘤与微生物之间相互作用的奥秘,并为癌症治疗提供新的策略。
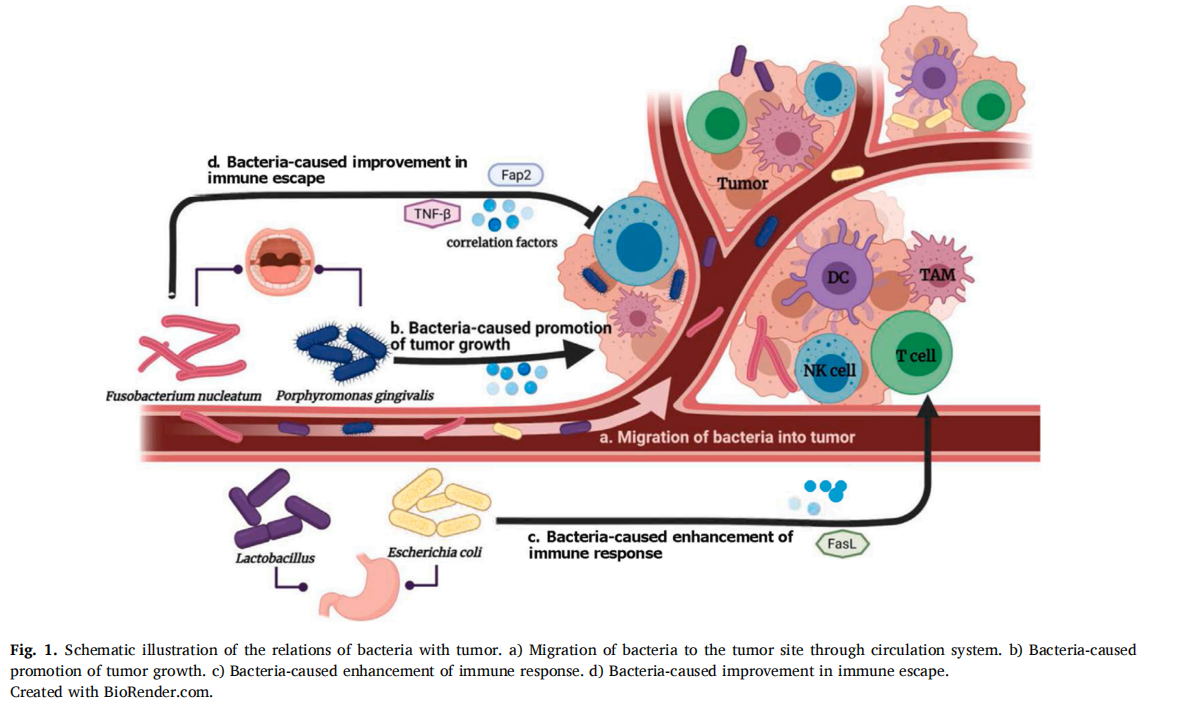
细菌与肿瘤的关系示意图
二 肿瘤微生物检测方法
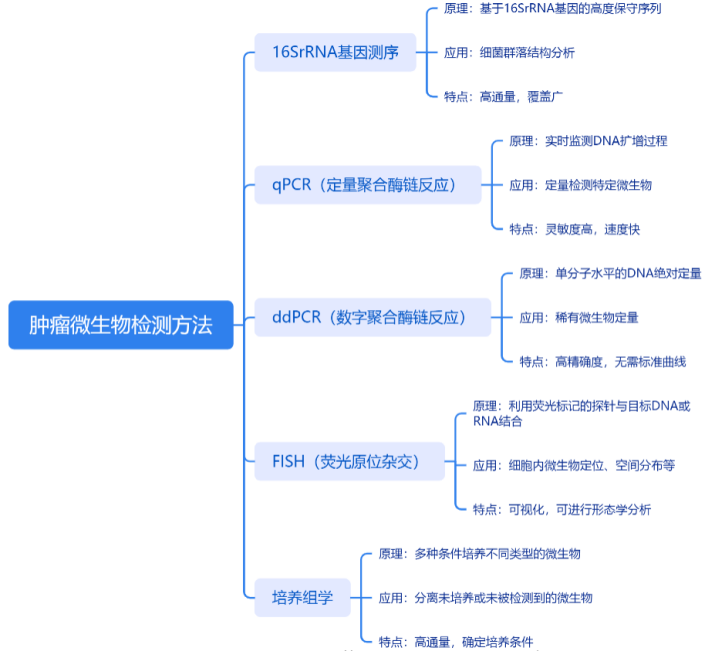
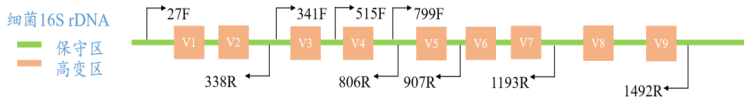
2.1 16S扩增子测序
通过16S rRNA基因的特定区域(如V3-V4区)进行PCR扩增,然后进行高通量测序,以鉴定样本中的微生物种类。能够揭示肿瘤组织中微生物的多样性、相对丰度和系统发育关系。
16S扩增子测序是鉴定细菌和古菌群落结构的常用方法,但通常分辨率限于属水平,且难以区分新物种。
实时荧光定量PCR(qPCR)技术是一种高度灵敏和特异的分子生物学检测手段,它通过实时监测PCR扩增过程中产生的荧光信号来定量分析DNA或RNA分子。
在肿瘤微生物qPCR检测中,该技术被用来定量分析肿瘤组织或相关样本中的微生物核酸,从而揭示微生物群落的组成和丰度,这对于理解肿瘤微环境中微生物的作用及其与肿瘤发展的关系具有重要意义。
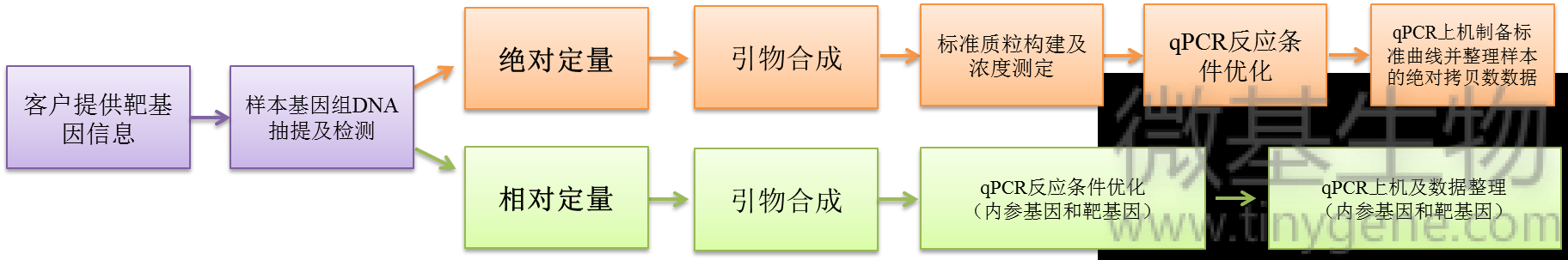
qPCR检测方法的优势:
Ø 高灵敏度和特异性:qPCR技术利用特异性引物和荧光标记的探针,能够检测到极少量的核酸模板,从而实现对微生物含量的精确测定。
Ø 快速性:qPCR实验通常在几小时内完成,从核酸提取到结果分析,大大缩短了检测时间。
Ø 可重复性和标准化:实验操作流程标准化,结果易于复制,便于不同实验室之间的结果比较。
Ø 定量能力:qPCR能够提供定量结果,不仅可以检测微生物是否存在,还可以测定其相对或绝对数量。
Ø 多样本并行检测:96孔或384孔的qPCR板可以同时检测多个样本,提高实验效率。
微基生物可以根据客户的目标和要求,设计针对目标菌株的特异性引物和探针,有效促进目标菌株的检测、筛选和鉴定。
2.3 肿瘤微生物ddPCR定量检测
肿瘤微生物的微滴式数字PCR(ddPCR)定量检测是一种高灵敏度和高准确度的核酸分子检测技术,它可以对肿瘤组织或体液中的微生物DNA进行绝对定量分析。而且较好的克服了肿瘤组织包埋标本DNA质量差、标本量有限等问题,目前已经被应用于多个研究领域,有望成为恶性肿瘤的诊断和监测的可靠工具。

ddPCR检测方法的优势:
Ø 高灵敏度和准确性:ddPCR能够检测到低至0.001%丰度的突变靶标,适合痕量微生物DNA的检测。
Ø 绝对定量:ddPCR不需要标准曲线,可以直接计算出目标DNA的拷贝数。
Ø 抗抑制剂能力:对PCR反应中的抑制剂具有较高的耐受性,适合直接从临床样本中进行检测。
Ø 无需依赖Ct值:与qPCR不同,ddPCR不依赖于Ct值,因此不受扩增效率的影响。
Ø 适合多重检测:可以同时检测多个靶标,提高实验效率。
微基生物提供ddPCR引物、探针设计、优化及检测服务,对微生物特异性种属精确定量。
2.4 种或属分类级别特异性的细菌16S FISH杂交检测
肿瘤微生物种或属分类级别特异性的细菌16S 荧光原位(fluorescence in situ hybridization,FISH)杂交检测是一种利用荧光标记的核酸探针针对肿瘤组织中微生物的16S rRNA基因序列进行特异性结合的分子生物学技术。这种技术可以对肿瘤组织中的微生物进行定性、定位和半定量分析,提供关于微生物种类、空间分布以及与宿主相互作用的详细信息。

16S FISH杂交检测的优势:
Ø 细胞定位能力:能够直接在组织切片中观察微生物的分布情况。
Ø 高特异性:利用特异性探针可以区分不同的微生物种类或属。
Ø 直观性:荧光显微镜下的直观图像便于分析和解释结果。
Ø 多标定量:可以同时使用多个探针,对多种微生物进行同时检测。
实验过程:
探针变性、标本变性、杂交、洗脱、杂交信号放大(生物素标记的探针)、复染、封片、荧光显微镜观察FISH结果。
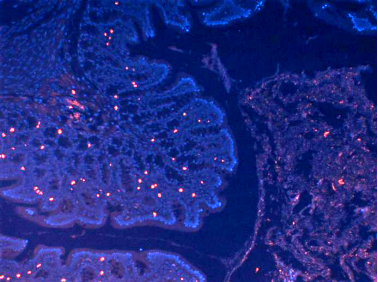
EUB338探针检测小鼠肠道中的所有细菌
微基生物提供微生物种属特异性16S FISH探针设计及检测服务,对种属特异性16S FISH探针系统性评测,确保其高特异性及通用性。
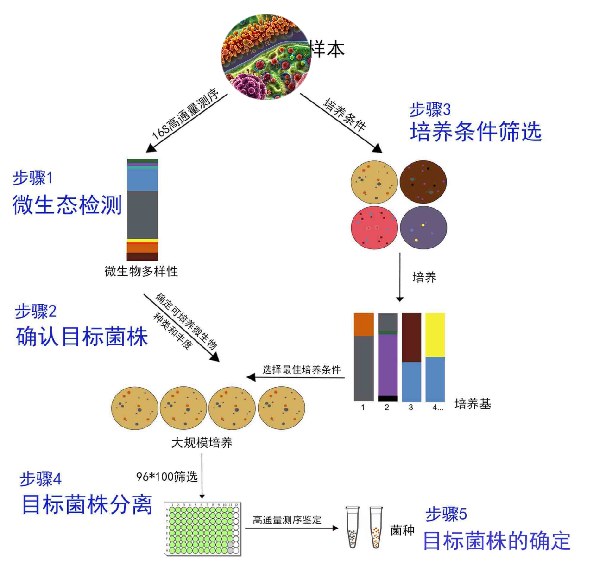
2.5 肿瘤组织中微生物的分离培养
培养组学(Culturomics)是一种综合方法,使用多种培养条件在不同培养基上培养细菌菌落,然后使用16S rDNA测序或MALDI-TOF质谱平台对其进行鉴定,是了解特定菌或菌群在宿主中作用的关键。可以较大程度的发现潜在的新菌株和微生物,丰富现有的可培养微生物资源库。
利用培养组学(多达48或96种条件)将原始样本中可培养微生物大大富集并进行微生物组学的检测,使原始样本中可检测的微生物类型大为丰富。为此后的微生物的培养分离验证了条件。
使用需氧/厌氧活菌保存液保存样本,微生物的种类更丰富,多样性更显著。
培养方法的优势
Ø 直接性:直接从肿瘤组织中分离微生物,避免了因样本处理导致微生物DNA污染的问题。
Ø 可重复性:培养条件标准化,实验结果可重复性强。
Ø 多样性分析:可以分析肿瘤组织中微生物的多样性和丰度。
Ø 功能研究:分离的微生物可以用于后续的功能研究,如药物敏感性测试和致病机制研究。
微基生物分离培养检测示意图
这些技术方法各有优势和局限性,通常结合使用以获得更全面的肿瘤微生物组信息。例如,16S扩增子测序可以提供微生物多样性的概览,而qPCR或ddPCR可以提供特定微生物的定量信息。FISH技术有助于理解微生物的空间分布,而培养分离则可以深入研究微生物的生物学特性。通过这些方法的综合应用,研究人员能够更好地理解肿瘤微生物组的组成、功能以及它们与肿瘤发展和治疗之间的相互作用。
三 样本类型
在多种实体肿瘤中检测到微生物群,包括肝脏、膀胱、肾脏、前列腺、胰腺、脑、食管癌、结肠癌、胃癌、肺癌、乳腺癌、口腔癌和妇科癌。肿瘤内微生物群已被证实与肿瘤发生、肿瘤进展和耐药有关。
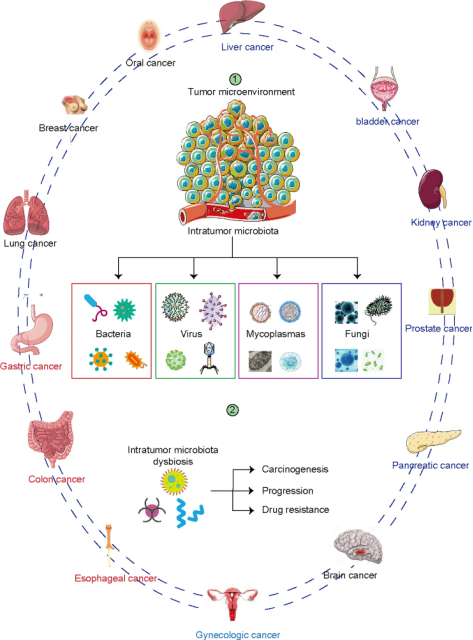
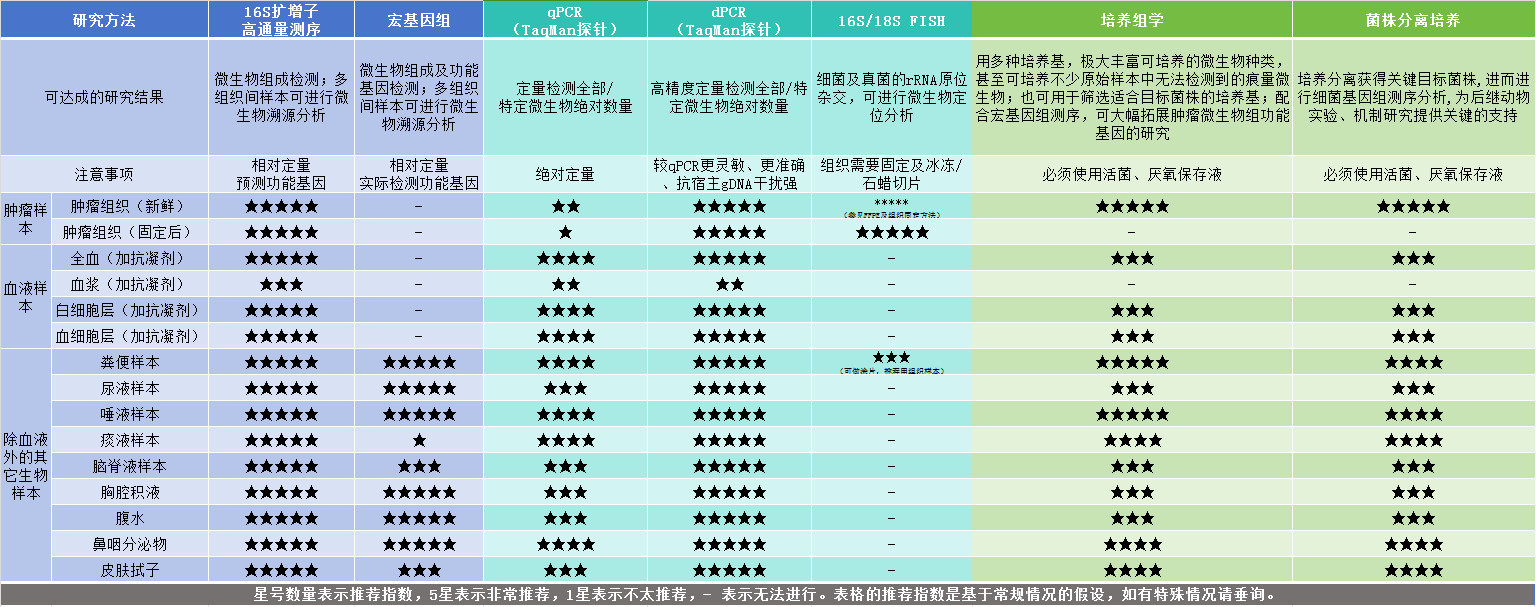
目前肿瘤微生物检测常用样本主要包括福尔马林固定石蜡包埋组织(formalin-fixed paraffin-embedding, FFPE)样本(包括手术切除和活检组织样本)和体液样本(包括外周血、脑脊液、浆膜腔积液上清液、唾液和尿液等),应依据临床需求、样本可及性及不同的检测平台性能选择合适的样本。
每种样本类型都有其特定的优势和局限性,选择合适的样本类型和检测方法对于准确评估肿瘤微生物组至关重要。具体样本详情请进一步咨询:021-50763698。
四 不同类型样本保存套装
实体肿瘤微生物样本种类繁多,可根据具体肿瘤样本的类型及选择的肿瘤微生物的检测平台选择合适的取样方式、取样量、保存方式等。
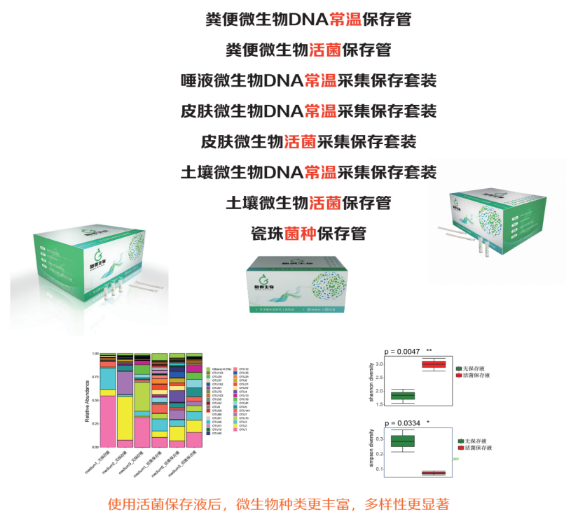
5.1 肝细胞癌的肿瘤微生物组特征: 16s+FISH+分离培养+qPCR

我们使用16S rRNA基因测序156个肝脏样本,分析了人类正常肝脏、肿瘤周围和肝细胞癌(Hepatocellular Carcinoma, HCC)组织的微生物谱,并确定了可能用于HCC诊断的特定微生物特征。我们还利用荧光原位杂交(FISH)和新鲜组织培养方法验证了HCC中肿瘤内微生物的存在。
5.1.1采用的检测方法
Ø 细菌16S rRNA基因全面DNA测序,发现肿瘤周围和HCC微生物群的α和β多样性均有所增加。
Ø 荧光原位杂交(FISH)显示,微生物DNA分布在肝细胞和红细胞的细胞质中。
Ø 通过新鲜组织培养在HCC组织中恢复了有活力的厌氧或好氧细菌。
Ø 采用独立队列(qPCR_cohort)对12份组织样本进行定量PCR验证。
5.1.2部分样本情况
FISH检测样本类型:手术过程中收集新鲜组织,制备石蜡切片,苏木精-伊红(H&E)染色,小心避免污染。新鲜组织被储存在无菌管中,在与患者身体分离后立即在冰上冷却。所有后续步骤均在冰上进行。
分离培养样本类型:将新鲜的肿瘤组织和肿瘤周围组织浸泡在无菌培养皿中的生理盐水中。
5.1.3结果展示
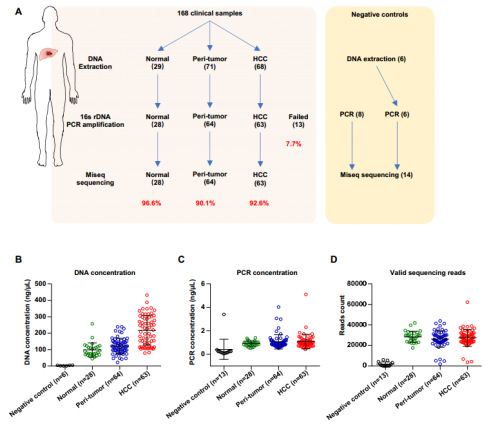
168个正常、肿瘤组织和HCC组织的16S rDNA测序
作者通过16S rDNA测序分析肝癌组织微生物群的结构和丰度,检测了68对HCC和癌旁组织配对样本、29例正常肝组织和3例独立的肝癌组织。与正常肝组织相比,HCC中微生物群的α多样性显著增加,癌旁组织富集有变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)和放线菌门(Actinobacteriota),而正常组织中副杆菌(Patescibacteria)和酸杆菌门(Acidobacteriota)丰度更高(图4A-D);而HCC组织中显示出更高的Proteobacteria、Firmicutes和Actinobacteriota,而Patescibacteria和Acidobacteriota的丰度较低(图4E-F)。以上结果表明,与对照组相比HCC组织和癌旁组织具有相似的微生物特征,但是分析其他菌群发现变形菌(Gammaproteobacteria)在HCC组织显著富集而不在癌旁组织中显著富集。为了验证这一结果,作者通过qPCR对Gammaproteobacteria进行分析发现与测序结果一致,癌组织中的Gammaproteobacteria丰度显著高于正常组织(图4I-J)。LEfSe结果证实,与健康对照相比HCC组织Proteobacteria、Firmicutes、Actinobacteriota增加,而Acidobacteriota、分歧杆菌(Parcubacteria)、酸杆菌(Acidobacteriae)减少(图5C-D);
正常、肿瘤周围和 HCC 组织中的肿瘤内微生物概况。(截取部分)
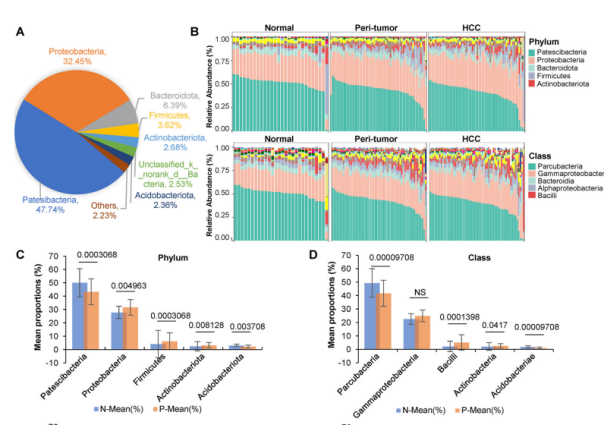
A.饼图显示了基于OTU的所有肝脏微生物群中每个门的比例。B.在NM,Pt和HccM的门和类水平的分类组成的条形图。
C和D.在门或类别水平上的丰度在正常微生物群和肿瘤周围微生物群之间有显著差异。
LEfSe分析揭示组织样本中的特定类群
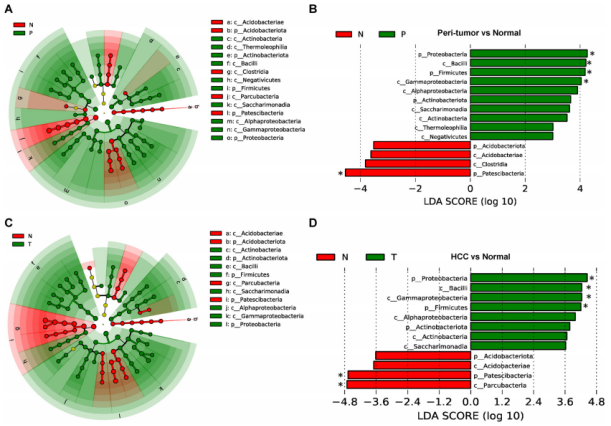
A.支序图显示正常(N)组织和肿瘤周围(P)组织之间具有显著差异丰度的分类树。B.显示正常人和肿瘤周围受试者之间细菌分类群的线性判别分析的直方图。C.支序图显示正常(N)组织和HCC(T)组织之间分类树具有显著差异的丰度。D.显示正常(N)和HCC受试者(T)之间具有显著差异丰度的细菌分类群的LDA评分。
肝癌的FISH分析 HCC组织的微生物的DNA富集于肝细胞和红细胞的细胞质中

A.将福尔马林固定的石蜡包埋(FFPE)肝癌切片脱石蜡,再水合。B.这两种探针最近都被用于分析人类癌症组织的肿瘤内微生物群。C.细胞核用二脒基苯基吲哚(DAPI)复染。单模染色图像。D.染色阴性。E.不同染色的合并图像。黄色箭头表示拥挤区域的红细胞聚集。箭头表示拥挤区域外的红细胞。F.苏木精-伊红连续组织切片染色。
为评估HCC中微生物DNA的分布和丰度,作者使用16S rRNA探针对癌组织及癌旁组织进行FISH实现,发现FISH信号分布于癌组织和癌旁组织,主要存在细胞质中;在癌旁组织中作者发现在释放最强信号的几个扩大的窦状区无核红细胞(RBC)中富集了FISH信号(红细胞在肝窦内的堆积表明受损肝组织严重充血)。
新鲜组织培养 HCC组织中存在活菌和感染性细菌
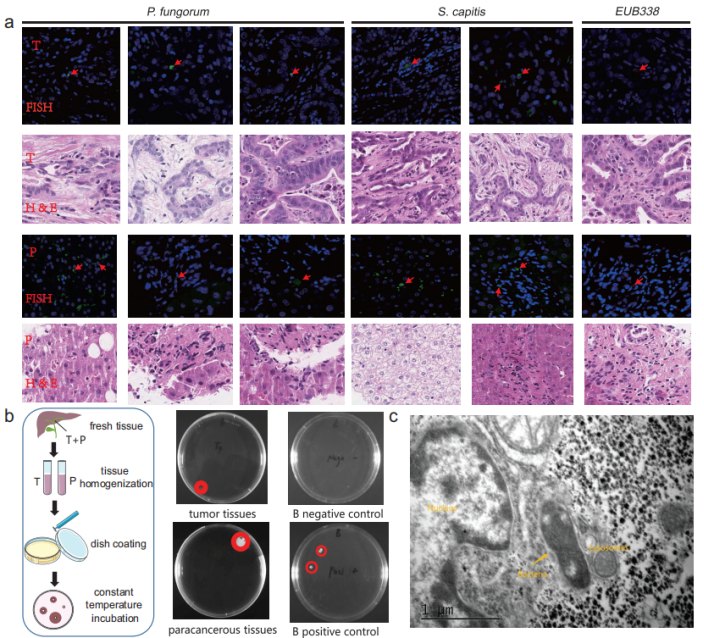
为研究HCC组织是否存在活菌,作者收集术后新鲜HCC组织通过组培法检测活菌的情况,作者通过设置阳性对照和阴性对照分别在好氧与厌氧环境中进行实验,发现阳性对照中环境样本在好氧培养下产生了数千个菌落,而厌氧培养产生了30+可见菌群,阴性对照没有产生可见菌,表明组培法的可靠性是比较高的。作者通过对培养出来的菌群进行菌落PCR和16S rDNA测序,共鉴定出13个菌落,其中2个菌落为金黄葡萄球菌(Staphylococcus aureus)、8个菌落为罗氏菌属(Rothia)兼性厌氧菌,2个菌落为芽胞杆菌(Bacillaceae)的好氧菌,1个是属于棒状杆菌(Corynebacterium sp.)的嗜氧微细菌。以上结果表明HCC组织中含有活的瘤内细菌。
qPCR 采用独立队列(qPCR_cohort)对12份组织样本进行定量PCR验证
为了验证16SrRNA测序的结果,我们使用来自16S_rRNA队列和包含12 个样本的独立队列(qPCR_cohort)的样本对Gammaproteobacteria进行qPCR分析。与16SrRNA基因测序的结果一致,癌组织中的Gammaproteobacteria丰度显着更高(Student’st检验,16S_rRNA队列中P= 0.033,qPCR_队列中P= 0.0005)高于正常样本。
qpcr_队列包含6例HCC癌和6例癌周标本,分别来自临床手术中7例男性和3例女性患者。
本文介绍了微生物群与HCC的关系,并探讨了与HCC进展相关的肿瘤内微生物群,发现HCC微生物群多样性增加。其中,链球菌科和乳球菌被认为是HCC肝硬化的标志。
5.2 肿瘤胞内菌促进乳腺癌的转移定植 16s+qPCR+FISH

该研究利用小鼠自发乳腺癌模型,通过qPCR和16S测序,发现肿瘤组织相对于环境对照和阴性对照有大量的微生物存在,并且肿瘤组织和正常组织的细菌来源类似,但是从“属”的水平来看,肿瘤组织有特定的微生物分布特征。这项研究揭示了肿瘤微生物群落在乳腺癌转移中的潜在作用,并为开发新的癌症治疗策略提供了科学依据。
5.2.1方案设计流程
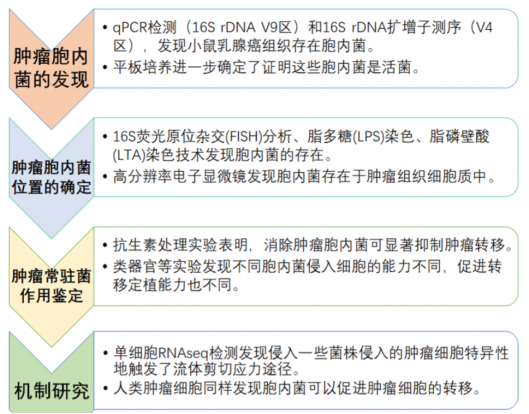
5.2.2采集类型
新鲜的正常乳腺组织、乳腺肿瘤组织和淋巴转移组织立即用无菌DMEM培养基转移到15ml的无菌锥形管中。样品在干净无菌的细胞培养罩中用高压灭菌的解剖工具进行处理。肿瘤组织切成1mm小块。
5.2.3结果展示
qPCR绝对定量及细菌培养 乳腺癌组织中存在活的胞内菌
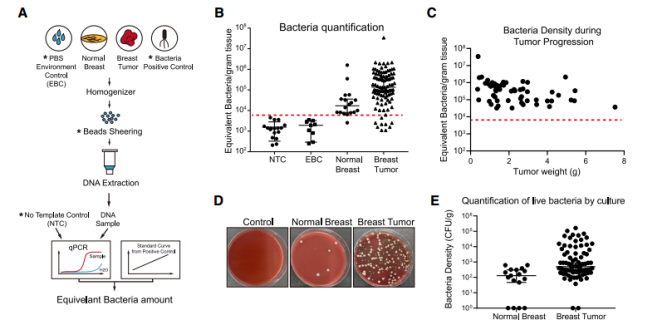
组织与自发乳腺癌(BT)模型小鼠肿瘤组织菌群的qPCR绝对定量及细菌培养分析
本研究在小鼠自发乳腺癌(BT)模型中构建了严格的肿瘤菌群研究体系。实验设置了4个分组:负对照(NTC)、环境背景对照(EBC)、小鼠正常乳腺细胞、小鼠乳腺肿瘤细胞。通过经过优化的Taqman qPCR检测(16S rDNA V9区),发现虽然正常乳腺细胞也含有一些胞内菌,但是乳腺癌组织的胞内菌的丰度有近10倍的显著增加。随着肿瘤体积的增大,细菌密度相对保持不变。通过平板培养,进一步确定了证明这些胞内菌是活菌,并且在乳腺癌组织中的细菌载量要显著多于正常组织。
16S扩增子测序 不同分组中胞内菌的多样性不同

胞内菌多样性分析
实验进一步通过16S扩增子测序来检测组织胞内菌的组成。由于组织的胞内菌生物量非常低,因此本研究优化了16S文库构建程序,包括对16S rDNA V4区进行检测,以及增加生物素富集步骤以减少非特异性基因组序列。最终获得了比常规肠道微生物检测方法灵敏度高得多的104当量细菌/g组织的可靠结果。结果发现,对照组(NTC和EBC)、正常乳腺组织和BT组织中在门和属水平具有不同的微生物群落组成。PCA结果也具有显著差异。阴性对照样品中的大多数微生物是变形菌门,而组织样品中富集了厚壁菌门。排除污染后,研究者发现与正常乳腺组织相比,BT组织的α多样性降低,表明某些微生物的选择和扩增。BT组织显示出厌氧菌的急剧减少和兼性厌氧菌的显著增加。以上结果表明,自发肿瘤组织中确实含有大量的活细菌。
16S荧光原位杂交(FISH)分析 大量肿瘤常驻菌位于胞质中
利用16S荧光原位杂交(FISH)分析、脂多糖(LPS)染色、脂磷壁酸(LTA)染色技术,以及高分辨率电子显微镜,研究者进一步发现在肿瘤组织细胞中有大量微生物的存在,而这些结果在通过抗生素处理实验中进一步得以验证。

大量常驻菌位于细胞质中
这项研究揭示了肿瘤微生物群落在乳腺癌转移中的潜在作用,并为开发新的癌症治疗策略提供了科学依据。
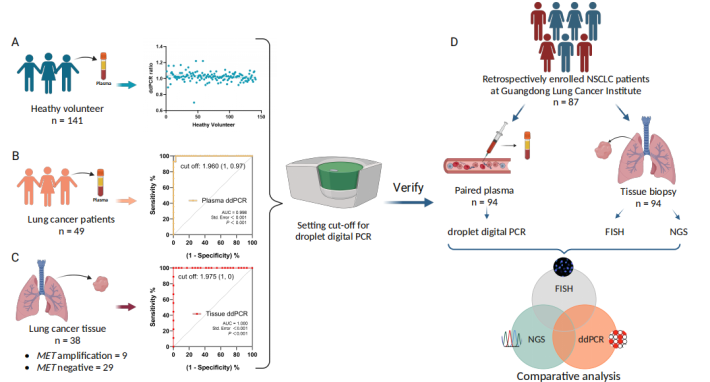
5.3 扩增在晚期非小细胞肺癌患者中:现实世界的比较研究16s+FISH+ddPCR

文章旨在评估血浆ddPCR和组织NGS在检测局部晚期或转移性NSCLC患者MET扩增的性能,并与组织样本的荧光原位杂交(FISH)技术进行了比较。血浆ddPCR与FISH具有较高的一致性(敏感性74.1%、特异性92.5%和准确度87.2%,kappa值为0.68),在MET扩增检测方面优于组织NGS(kappa值为0.64)。血浆ddPCR和组织NGS联合检测与FISH具有较高的一致性(敏感性92.3%、特异性89.2%和准确度90.1%,kappa值为0.77)。对于FISH和血浆ddPCR检测到MET扩增并接受MET-TKI治疗的NSCLC患者,其疗效相当。
5.3.1采用的检测方法:
Ø FISH:作为检测MET扩增的金标准,用于评估肿瘤组织样本中的基因拷贝数(GCN)和MET与染色体7中心粒(MET/CEP7)的比率。
Ø ddPCR:一种先进的技术,用于绝对定量核酸分子,无需标准曲线。它通过将反应分成包含单个核酸模板分子的数千个微滴来检测基因突变或表达丰度,具有0.1%的灵敏度。
Ø 组织NGS:评估MET扩增,通过计算与正常MET状态样本池建立的基线相对的GCN比率。
5.3.2 样本类型
所有患者通过肿瘤组织活检使用FISH进行MET扩增检测,并在活检前后约2周收集成对血浆样本。如果有肿瘤组织,则进行组织NGS。
ddPCR检测:组织样本的gDNA约为12ng,外周血样本的cfDNA至少为1.2ng
5.3.3结果展示
设置 ddPCR 的截止值
为了确定血浆ddPCR 的截止值,我们首先验证了其在健康个体中的一致性。我们分析了141份接受常规医学检查的志愿者外周血样本,发现ddPCR比值均匀分布在1.0和1.2之间,平均在1左右。然后我们分析38份肺癌组织和49份已确认 FISH 结果的肺癌患者血浆。发现MET阳性组织血浆和组织ddPCR比值均超过2,而MET阴性组织血浆和组织ddPCR比值低于2。得到的ROC曲线的曲线下面积(AUC)分别为血浆和组织的0.998和1.00,因此我们将ddPCR检测MET扩增的截止值设为2.0。
FISH和ddPCR MET扩增诊断性能一致性分析
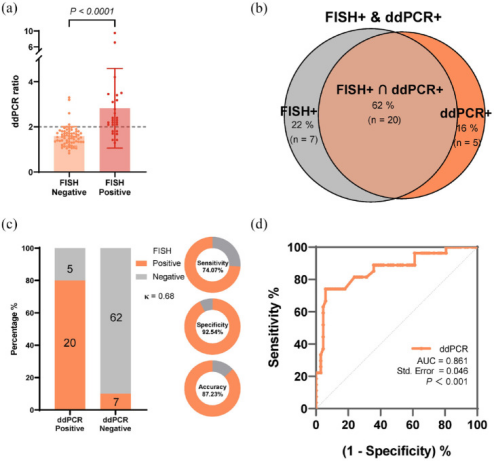
(a)MET-FISH阳性组和阴性组ddPCR比率的小提琴曲线图。(b)MET-FISH阳性组和MET-ddPCR阳性组的维恩图。
(b)ddPCR阳性和FISH阳性样本之间的总体相关性。(d)血浆ddPCR的ROC曲线分析。
AUC曲线下面积;ddPCR液滴数字聚合酶链反应;FISH荧光原位杂交;MET间质-上皮转化;ROC受试者工作特征。
FISH 是检测MET扩增的标准方法。本研究基于94份配对样本,评估了血浆 ddPCR与FISH检测MET扩增的一致性。不同临床分期和肿瘤负荷的血浆ddPCR MET扩增检测无显著差异。我们的分析显示 FISH 阴性组和阳性组的血浆ddPCR比率存在显著差异,平均比率分别为1.59和2.82 图2(a)血浆ddPCR阳性(n=25) 和FISH阳性(n=27)重叠率为62%图2(b)5份FISH阴性样本经血浆 ddPCR 检测呈阳性,7份FISH 阳性样本经血浆 ddPCR检测呈阴性,灵敏度、特异性、准确度和kappa值分别为74.1%、92.5%、87.2% 和0.68图2(c)此外,ROC曲线显示血浆ddPCR检测MET扩增的AUC为0.861 图2(d)。这些发现表明血浆ddPCR和FISH在检测MET扩增方面具有相当的一致性。
于ddPCR方法的MET FISH 扩增的诊断性能
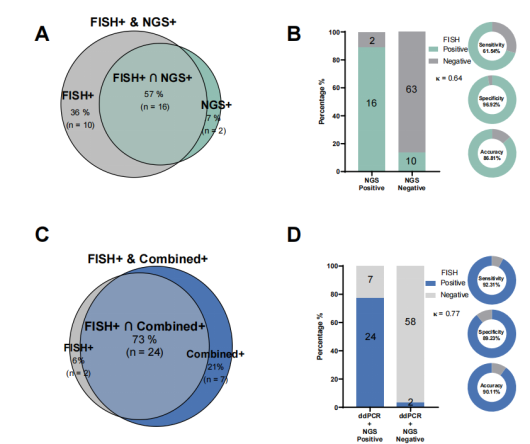
由于肿瘤组织量不足,3例患者无法进行NGS分析,其中1例为FISH阳性,另2例为FISH阴性。其余91例(96.8%)同时获得了FISH和NGS结果。与血浆ddPCR结果相似,组织NGS阳性(n=18)和FISH阳性(n=26)也较常见(57%)[补充图S2(A)]。2例FISH阴性病例为组织NGS阳性,10例FISH阳性病例为组织 NGS 阴性。因此,组织NGS检测MET扩增的灵敏度、特异性、准确度和κ值分别为61.5%、96.9%、86.8% 和0.64[补充图 S2(B)]。虽然组织NGS与FISH的MET扩增一致率高于我们中心之前报告的值(62.5%),但血浆ddPCR方法在数值上表现出了更优异的性能。
这项研究支持了血浆ddPCR在检测晚期NSCLC患者MET扩增中的效用和可行性,并指出了进一步验证这些发现的前瞻性临床试验的必要性。
5.4 人类肝细胞癌和邻近非肿瘤组织的微生物概况概述: 16S+FISH

肿瘤内微生物群落最近被发现存在于多种癌症中,并被发现复杂地参与肿瘤进展。因此,研究肝细胞癌(HCC)中肿瘤内微生物分布的特征和功能是必要的。
5.4.1采用的检测方法:
Ø 对HCC组织进行了荧光原位杂交(FISH),从而验证微生物在HCC中的存在。
Ø 使用16S rRNA测序技术对99个临床收集的肝癌和相邻非癌组织进行MiSeq测序,得到了两者内部微生物的全面图谱。发现肝癌组织中的微生物群落多样性(包括α和β多样性)显著高于癌旁组织。
5.4.2样本类型
肝癌及癌旁组织标本。所有肿瘤组织及邻近组织均行手术切除。
5.4.3结果展示
荧光原位杂交(FISH)证实了肝细胞癌中存在细菌
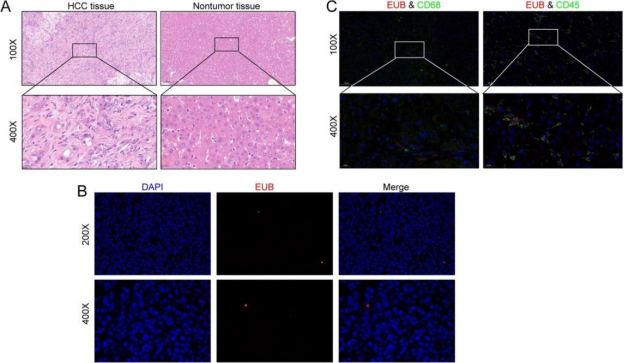
A.肝细胞癌组织和癌旁组织的H&E染色。 评估HCC和非肿瘤组织的典型特征B.细菌16S rRNA序列的B-FISH分析证实了细菌在HCC中的存在。HCC组织中含有细菌,尽管数量较少。C.代表性免疫细胞染色。CD68+细胞和CD45+细胞中均有细菌。其中CD68+细胞被认为是巨噬细胞。
16S rRNA测序 肝细胞癌和癌旁组织微生物群组成的差异
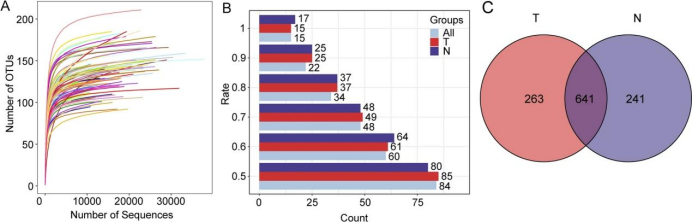
A.序列数据的稀疏度曲线。在特定的测序深度下,曲线趋于平缓,即数据量的增加产生的新otu相对较少,说明我们的测序数据深度是合理的。B. HCC和癌旁组织中不同样品覆盖率的核心微生物。在不同的样品覆盖水平下,从HCC和癌旁组织中获得的样品中共享微生物的数量是一致的。在所有样品中鉴定出15种核心微生物,并且在HCC和癌旁组织中一致。C.组间OTU数的集合分布分析。肿瘤组织中有263个独特的otu,邻近组织中有超过241个独特的otu,这表明两组之间的微生物种类存在显著差异。
肝癌和癌旁组织中微生物的Alpha 和Beta多样性
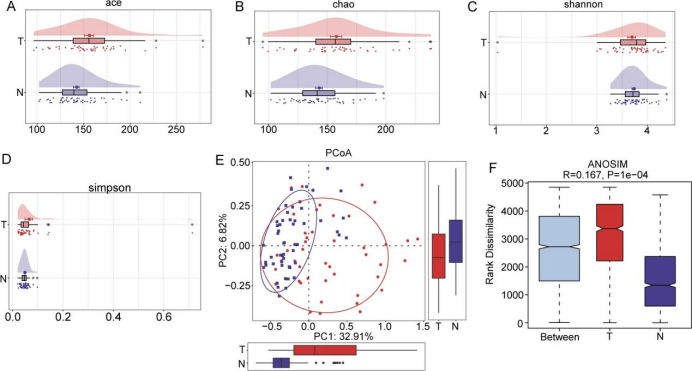
A组间Ace指数的差异。B组间Chao指数的差异。C组间 Shannon指数的差异。D组间Simpson指数的差异。E基于未加权UniFrac距离的细菌Beta多样性 PCoA。F基于ANOSIM 的微生物群Beta多样性
以上结果表明HCC和癌旁组织在单个样本的细菌丰富度和两组的整体组成上存在显著差异。
肝癌和癌旁组织微生物组成的差异
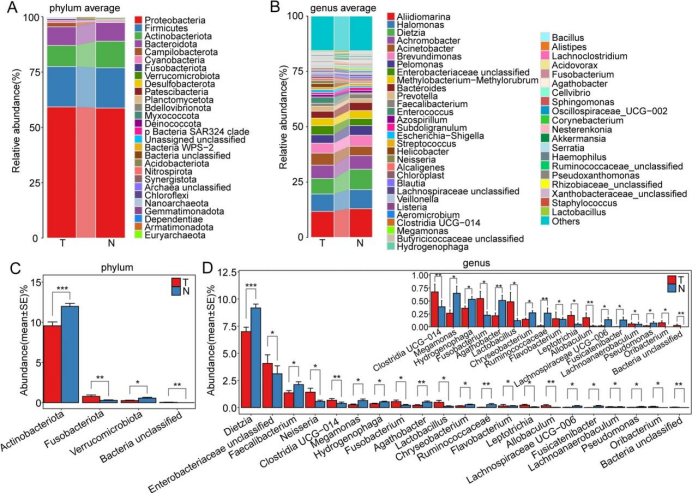
A.肝癌和癌旁组织在门水平上的微生物组成。B属水平上肝癌和癌旁组织的微生物组成。C基于Mann-Whitney U检验的研究组在门水平上的微生物差异。D .基于Mann-Whitney U检验的研究群体在属水平上的微生物差异
基于LEfSe和随机森林识别肝细胞癌与癌旁组织之间的微生物组及其功能的差异
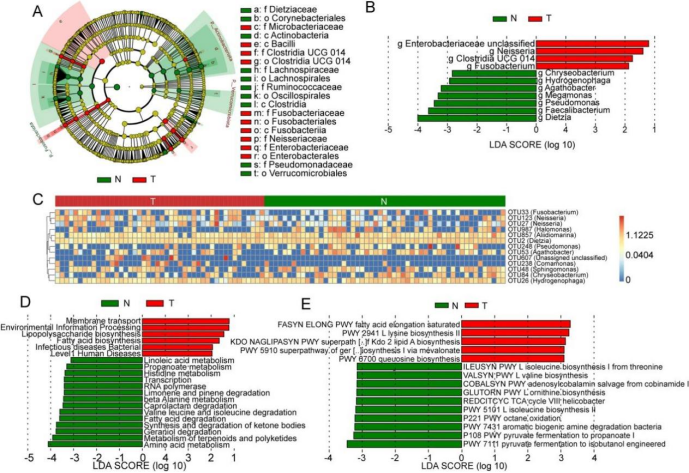
A.基于LEfSe和随机森林的HCC和癌旁组织间微生物组及其功能的差异包含从门到属水平的不同细菌分类群的进化图谱。B.属水平微生物群差异LDA直方图。C.根据随机森林算法确定的对群体差异贡献最大的微生物。D .基于KEGG数据库的组间代谢途径差异。E .基于MetaCyc数据库的组间代谢途径差异。
文章综合评价了HCC组织的瘤内微生物图谱,初步探讨了微生物群落参与脂质代谢变化及影响HCC进展的作用机制,为该领域的进一步研究奠定了基础。
5.5 利用培养组学技术分离培养肺部微生物群研究 16S+培养组学
使用培养组学技术分离培养肺部微生物群,初步了解人体肺部可培养细菌的组成特点,建立呼吸道微生物菌库,为以后进行重点菌株的功能研究提供菌株条件。
培养组学工作流程示意图
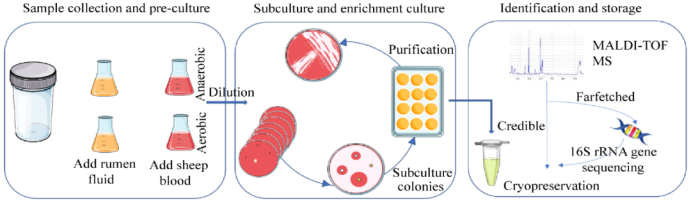
针对6个临床肺泡灌洗液样本,使用补充不同添加剂的血培养瓶对样本进行预增菌,在预增菌不同的时间点对血培养瓶内细菌进行分离培养和保藏,使用MALDI-TOF质谱和16S rRNA基因测序鉴定分离菌株。
共获得101种已鉴定细菌,6株潜在新菌,2株真菌。其中细菌包括5个门、14个纲、24个目、35个科和45个属。预增菌前期的时间点分离菌种数较多,厌氧环境分离菌种多于需氧条件,在预增菌时添加羊血和瘤胃液起到良好的分离效果,本实验分离的肺部微生物群与人体其他部位(尤其是肠道)有很大的交叉。
肺部微生物群分离培养情况
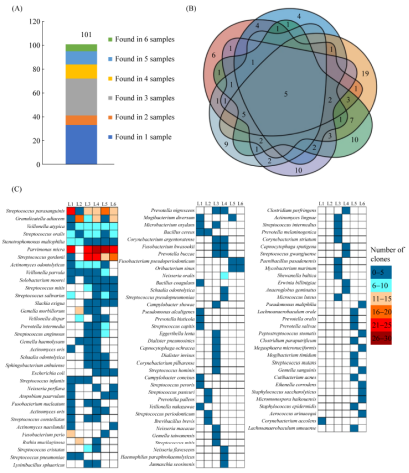
6个样本中分离细菌总体情况 A: 6个样品中分离细菌的数量;B:不同肺泡灌洗液样本间分离细菌的维恩;C:从6个样品中分离出的细菌及其在每个样品中的分布。
利用培养组学技术可以实现对肺部微生物群的分离培养,继续探索对肺部微生物群培养的优化方法具有现实意义。
微基生物科技(上海)有限公司,是专门从事微生物生态学/微生态相关研究的高科技服务公司。致力于为客户提供微生态整体研究服务,欢迎有肿瘤微生物研究需求的老师们前来咨询了解。
参考文献:
1. Bacteria and tumor: Understanding the roles of bacteria in tumor genesis and immunology
2. Intratumor microbiome in cancer progression: current developments, challenges and future trends.
3. The Intratumoral Bacterial Metataxonomic Signature of Hepatocellular Carcinoma.
4. Tumor-resident intracellular microbiota promotes metastatic colonization in breast cancer .
5. Plasma ddPCR for the detection of MET amplification in advanced NSCLC patientsa comparative real-world study.
6. Overview of microbial profles in human hepatocellular carcinoma and adjacent nontumor tissues.
7. 利用培养组学技术分离培养肺部微生物群研究。

 微基生物 您自己的微生态研究团队|专注微生态研究与应用
微基生物 您自己的微生态研究团队|专注微生态研究与应用





